Reacción en cadena de la polimerasa: qué es la PCR más allá de su uso por la COVID-19
La reacción en cadena de la polimerasa, abreviada PCR por su nombre en inglés (Polymerase Chain Reaction) se ha popularizado en el lenguaje coloquial a raíz de la pandemia de COVID-19, pero su uso en laboratorio es muy habitual desde hace muchos años, para usos que van desde la secuenciación del ADN hasta la generación […]

Última modificación: 13 mayo 2021
La reacción en cadena de la polimerasa, abreviada PCR por su nombre en inglés (Polymerase Chain Reaction) se ha popularizado en el lenguaje coloquial a raíz de la pandemia de COVID-19, pero su uso en laboratorio es muy habitual desde hace muchos años, para usos que van desde la secuenciación del ADN hasta la generación de perfiles de ADN forense a partir de muestras genéticas, pasando por la identificación de patógenos gracias a la detección de la ausencia o presencia de sus genes.
En este post explicaremos en qué consiste la reacción en cadena de la polimerasa, incluyendo sus principios y sus etapas, y para qué se usa, aparte de detectar la presencia del gen (ARN en este caso) del coronavirus SARS-CoV-2, que causa la COVID-19.
Una emulación de una actividad celular natural
La reacción en cadena de la polimerasa (PCR) nace de la dificultad de estudiar trozos de ADN aislados en análisis moleculares y genéticos que requieren cantidades significativas de material genético. La PCR permite “amplificar” o “fotocopiar” diminutos segmentos de ADN millones de veces, por lo que a menudo se habla de está técnica como uno de los avances científicos más importantes en biología molecular, que valió a su creador, Kary B. Mullis, el Premio Nobel de Química en 1993.
La PCR se basa en una actividad enzimática que en las células de nuestro organismo se produce de forma natural: las ADN polimerasas pueden obtener dos copias idénticas de las cadenas de ADN nuclear, que en la mitosis repartirán a las células hijas. De la misma manera, en está técnica las polimerasas (generalmente las denominadas Taq) podrán replicar, como una fotocopiadora, un fragmento de ADN, en varios ciclos (entre 20 y 35), que son cambios repetidos de temperatura, para obtener una gran cantidad de copias para poder analizar, en una reacción en cadena a la que debe su nombre.
Usos de la reacción en cadena de la polimerasa
Una vez amplificado, el ADN obtenido por PCR se puede usar en muchos procedimientos de laboratorio diferentes. Por ejemplo, la mayor parte de técnicas de mapeo del Proyecto del Genoma Humano se basaban en la PCR.
Además, también se usa para un sinfín de técnicas clínicas y de laboratorio habituales, incluidas las huellas digitales de ADN (pruebas de paternidad o análisis forenses, por ejemplo), detección de microorganismos (bacterias como Salmonella o Listeria en agua o alimentos, Legionella o agua, etc., virus como el sida, la hepatitis o el COVID-19) y el diagnóstico de trastornos genéticos.
Ventajas de la reacción en cadena de la polimerasa
La reacción en cadena de la polimerasa tiene algunas ventajas significativas respecto a los tradicionales cultivos, que son el método que tradicionalmente asociamos a la microbiología: placas de petri y tubos de ensayo colocados en estanterías, microscopio, colocación de los tejidos en placas de cultivo que se introducen en una incubadora y esperar a ver qué crece, de forma lenta pero segura. A menudo este proceso implica ensayo-error, descartando especie tras especie hasta que solo queda una, a través de la observación de sus características y su comportamiento. Por ejemplo, la E. coli y la Pseudomonas aeruginosa tienen células gramnegativas en forma de bastoncillo, por lo que observadas con microscopio se ven similares, pero sólo mediante un cultivo se puede comprobar que la E. coli metaboliza la lactosa.
Sin embargo, las pruebas moleculares como la PCR han aportado algunas ventajas a las pruebas de cultivo. Las principales son:
- Rapidez en la respuesta: reacción en cadena de la polimerasa tarda tan sólo unas horas en arrojar resultados, mientras que las pruebas de cultivo bacteriano pueden alargarse días.
- Sensibilidad: hay organismos que son casi imposibles de cultivar en un laboratorio, que la PCR permite detectar en unos niveles bajos. Esto ayuda a reducir falsos negativos, ya que puede darse la posibilidad de que una muestra positiva se de por negativa porque no se dispone de cantidad suficiente para analizarla.
Qué se necesita para llevar a cabo una PCR
Una plantilla de ADN: en primer lugar se necesita el ADN que se debe copiar, que generalmente se extrae y se purifica de la sangre o de otro tejido.
Cebadores o iniciadores (primers, en inglés): se trata de oligonucleótidos monocatenarios que se unen a la plantilla de ADN por los extremos y sirven para que la polimerasa inicie la reacción para empezar a sintetizar el material genético. Se necesitan dos cebadores para la PCR, cada uno de ellos complementario a una cadena de ADN que queremos amplificar, y delimitan la zona del ADN a amplificar, es decir, que corresponden a:
- Una ADN polimerasa (por ejemplo, Taq polimerasa o Pfu): para sintetizar el ADN como hemos explicado.
- dNTP (Trifosfatos de desoxirribonucleótidos): los componentes básicos del ADN son 4 tipos de nucleótidos, integrados por 4 bases nitrogenadas (adenina, guanina, citosina y timina), por lo que para poder obtener nuevas moléculas de ADN (las copias) son indispensables estos componentes.
- Una solución tampón: se trata de la creación de un entorno óptimo para que tenga lugar la reacción en cadena, en el que poder regular el pH, es decir, las condiciones de acidez o basicidad en las que se produce la síntesis del material genético.
- Solución de sal de cloruro de magnesio: el cloruro de magnesio se disocia y se libera magnesio, cuyos iones de carga positiva (+2) se usan como cofactores de la polimerasa.
Además de todos estos ingredientes, la reacción en cadena de la polimerasa implica un proceso de cambios de temperatura (los ciclos) que tienen lugar en un equipo llamado termociclador, que se programa para alterar la temperatura de la reacción cada pocos minutos para permitir la desnaturalización y síntesis del ADN, de manera que el proceso completo finalice en unas pocas horas.
Etapas de la PCR
1. Desnaturalización del ADN
Es el momento en el que la plantilla de ADN de doble cadena se calienta para separarlo en dos cadenas simples.
¿Qué ocurre en esta etapa?
- El conjunto de componentes de la PCR, la plantilla de ADN y el resto, se calienta a 94-95ºC.
- Durante esta etapa, el cóctel que contiene la plantilla de ADN y todos los demás ingredientes centrales se calienta a 94-95°C.
- La alta temperatura hace que los enlaces de hidrógeno entre las bases en dos cadenas de la plantilla de ADN se rompan y las dos cadenas se separen.
- Estas dos cadenas individuales de ADN actuarán como plantillas para la producción de las nuevas cadenas de ADN (las copias).
- En esta etapa la temperatura debe mantenerse un tiempo suficiente que garantice que las dos cadenas se hayan separado por completo.
- Esta parte suele tardar entre 15 y 30 segundos.
2. Alineación del ADN
Durante esta etapa, se baja la temperatura para permitir que los cebadores (primers) de ADN se adhieran a la plantilla de ADN.
¿Qué ocurre en esta etapa?
- Durante esta etapa, la reacción en cadena baja su temperatura a 50-65°C. Esto permite que los primers o cebadores se adhieran a un lugar concreto específico en la plantilla de ADN monocatenario (de una sola cadena) mediante enlaces de hidrógeno (la temperatura exacta depende de la temperatura de fusión de los primers que se utilicen).
- Los primers o cebadores son secuencias de ADN que poseen de 20 a 30 bases de longitud, diseñados para complementar secciones cortas de este ADN en cada extremo de la secuencia a copiar.
- Estos primers sirven como punto de partida para sintetizar el ADN, ya que la polimerasa solamente puede añadir bases de ADN a una doble hebra, es decir, que sólo cuando el primer se ha unido a la secuencia, la polimerasa puede unirse y empezar a generar la nueva hebra complementaria de ADN, a partir de bases sueltas.
- Estas dos hebras de ADN separadas son complementarias, y van en sentido opuesto (desde un extremo, el extremo 5 ‘, al otro, el extremo 3’); de manera que hay dos primers: uno directo y uno inverso.
- Este paso suele tardar entre 10 y 30 segundos.
3. Ampliación del ADN
Es el paso final, la parte en la que la temperatura aumenta y la nueva cadena de ADN es producida por la enzima polimerasa Taq.
¿Qué ocurre en esta etapa?
- El calor se incrementa a 72ºC para permitir que el nuevo.
- La ADN polimerasa Taq proviene de bacterias procedentes generalmente de aguas termales que pueden llegar a temperaturas superiores a los 90ºC, es decir que viven muy bien en ambientes cálidos, por lo que que puede soportar las temperaturas necesarias para romper las hebras de ADN en la etapa de desnaturalización del ADN. No sería posible, por ejemplo, con ADN polimerasa de la mayoría de los demás organismos (en los humanos, por ejemplo, funciona a temperatura corporal, unos 37ºC, y no soporta temperaturas más altas).
- 72°C es, por lo tanto, la temperatura ideal para que la polimerasa Taq construya la hebra complementaria. Se adhiere al primer y luego agrega bases de ADN a la hebra individual una por una en la dirección de 5 ‘a 3’.
- Como resultado obtendremos una nueva cadena de ADN y una molécula de ADN de doble cadena.
- Este paso puede tardar alrededor de un minuto (para copiar 1.000 bases de ADN), aunque esto depende de la longitud de la secuencia de ADN que se va a amplificar.
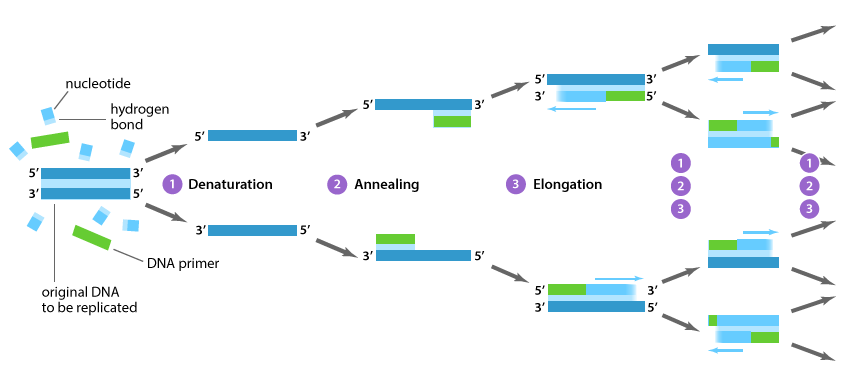
Repeticiones
La reacción en cadena de la polimerasa requiere repetir estos tres procesos o ciclos térmicos de 20 a 40 veces, duplicando el número de copias de ADN cada vez.
¿Qué ocurre en cada repetición?
- Los nuevos fragmentos de ADN que se generan durante la PCR a su vez sirven como plantillas a las que la polimerasa puede unirse y comenzar a generar más ADN.
- El resultado es un gran número de copias de un segmento de ADN concreto obtenidas en un periodo de tiempo mucho más corto que otro tipo de técnicas, unas pocas horas o incluso menos si se trata de una PCR con máquinas de alta velocidad.
Tipos de reacción en cadena de la polimerasa
Hoy en día existen numerosas variaciones de la reacción en cadena de la polimerasa, según el objetivo y los requisitos que se tenga. Las más habituales son:
PCR anidada
Está variación de la reacción en cadena de la polimerasa se usa cuando se quiere incrementar la sensibilidad y la especificidad de la detección. Por eso se duplica el paso de la amplificación con distintos pares de primers en cada uno. Primero se lleva a cabo una reacción en cadena con los cebadores externos para amplificar la parte más extensa de ADN, que contiene el segmento que tenemos por objetivo, y luego está amplificación se usa como ADN plantilla de una segunda PCR con cebadores internos para copiar ese segmento específico.
PCR in situ
Se suele usar para detectar cadenas de ADN dentro de células que con otras técnicas no se pueden detectar, por lo que se hace en tejidos. En contrapartida tiene menor capacidad de amplificación.
PCR múltiple
En esta variante se usan varias parejas de cebadores en una misma reacción en cadena para varias ampliaciones. Cada pareja corresponde a un virus distinto, y así se puede detectar una misma reacción de varios patógenos a la vez, como por ejemplo diferentes virus respiratorios.
RT-PCR (con transcriptasa inversa)
Es la que se lleva a cabo en el diagnóstico de virus como el SARS-CoV-2 que causa la COVID-19. Este tipo de PCR es necesario para virus que tienen genoma de ARN, que requieren un paso adicional en el proceso: después de aislar y purificar el ARN, se usa la transcriptasa inversa (llamada también RT, por sus siglas en inglés, Reverse Transcription) para sintetizar una molécula de ADN complementario que sirve de inicio para la PCR convencional.
Consta, por lo tanto, de tres pasos:
- Primer paso: retrotranscripción a partir del ARN.
- Segundo paso: amplificación a partir de la primera hebra de ADNc.
- Tercero paso: PCR estándar.
PCR en tiempo real o cuantitativa
La ventaja principal de esta PCR es que permite monitorizar (medir) en tiempo real la cantidad de fragmentos de material genético que se van produciendo, es decir, durante cada ciclo de amplificación, y no al final, sin sacrificar la precisión. Se requieren reactivos fluorescentes y un termociclador que mide la fluorescencia en un momento concreto de cada ciclo de amplificación. Esto es especialmente interesante si lo que se desea testar es un microorganismo en alimentos muy perecederos o de vida útil corta.
PRC de células viables.
Como paso previo a la amplificación del fragmento de ADN diana, la muestra se somete a una neutralización del material genético “libre” en la suspensión, o el procedente de células dañadas o muertas. Con este paso inicial, los resultados de la PRC evidenciarán solamente la presencia de células vivas presentes en nuestra muestra.
Aconsa, laboratorio especializado en PCR alimentarias y de aguas
Si requieres una prueba de diagnóstico en laboratorio como la reacción en cadena de la polimerasa, contáctanos y te asesoramos. Podemos ofrecerte PCR en tiempo real para la detección de Legionella en todo tipo de aguas. Además nuestra técnica nos permite identificar exclusivamente las células de Legionella que están vivas, y emitimos el resultado cuantificando las unidades genómicas detectadas, y su correlación con las ufc/l. De este modo, nos alineamos con las normas UNE 100030 y el RD865/2003. Puedes acceder al ‘paper’ vía subscripción donde describimos esta tecnología pinchando aquí.


